基因拷贝数变异(gene CNV)是种群适应性进化的主要遗传基础之一,但传统检测种群间基因CNV分化方法存在不少难题。二代测序技术虽然成本较低,但其传统检测手段在面对CNV时,容易受到GC含量偏差、重复序列以及低测序深度的影响,通常需要进行较深的测序,在样本较多情况下无疑增加了成本。而三代测序技术虽然在CNV检测的准确度上表现较好,但费用相对较高。
最近,中国科学院武汉植物园研发了一种名为“ST4gCNV”的新方法。该方法通过MEGAHIT将短读长二代测序数据快速组装成contigs,模拟标准化的reads深度(10×)后再比对参考基因组,有效减少了技术噪声(图1)。另外,ST4gCNV聚焦于基因编码区的CNV可以有效避开低复杂度和重复基因组区域在识别基因的CNV时所带来的挑战。即使在低至10×的测序深度下,该方法也能可靠地检测出基因特异性的CNV。该流程提供了低预算且高效的CNV检测方案,助力进化生物学、保育遗传学和分子育种等研究。
该研究将ST4gCNV流程应用于莲属植物(莲Nelumbo nucifera、美洲黄莲N. lutea)的群体基因组分析,揭示了大量基因在两近缘种间存在显著拷贝数差异,其中花青素途径基因的CNV分化尤为突出,可能与花瓣颜色分化相关。同时,基因拷贝数的增加主要通过正向剂量效应影响物种间的基因表达差异,表现为一个物种中基因拷贝数较高的基因,其总表达量也随之增加。此外,莲的温带与热带型之间也有少量基因存在CNV的分化,且功能富集于催化活性和环境响应通路。这表明基因CNV在近缘物种分化和生态型适应进化中具有重要作用。在基准测试中,在较低测序深度下,ST4gCNV相较于现有的CNVkit工具表现更佳,特别是在检测具有序列分化的基因拷贝时,其灵敏度更高。此外,研究还发现,ST4gCNV预测的CNV区域伴随着更高的杂合度(主要由旁系序列变异引起)和转座子比例,这与CNV的典型特征相符,进一步验证了该方法的可靠性。
该研究以“A Strategy of Assessing Gene Copy Number Differentiation Between Populations Using Ultra-Fast De Novo Assembly of Next-Generation Sequencing Data”为题发表于分子生态领域经典期刊《Molecular Ecology Resources》。中国科学院武汉植物园被子植物早期演化学科组石涛研究员为论文的第一作者/共同通讯,Duke University的Mark D. Rausher教授、武汉植物园水生植物生物地理学学科组陈进明研究员为共同通讯作者。该研究受到国家自然科学基金项目等资助。
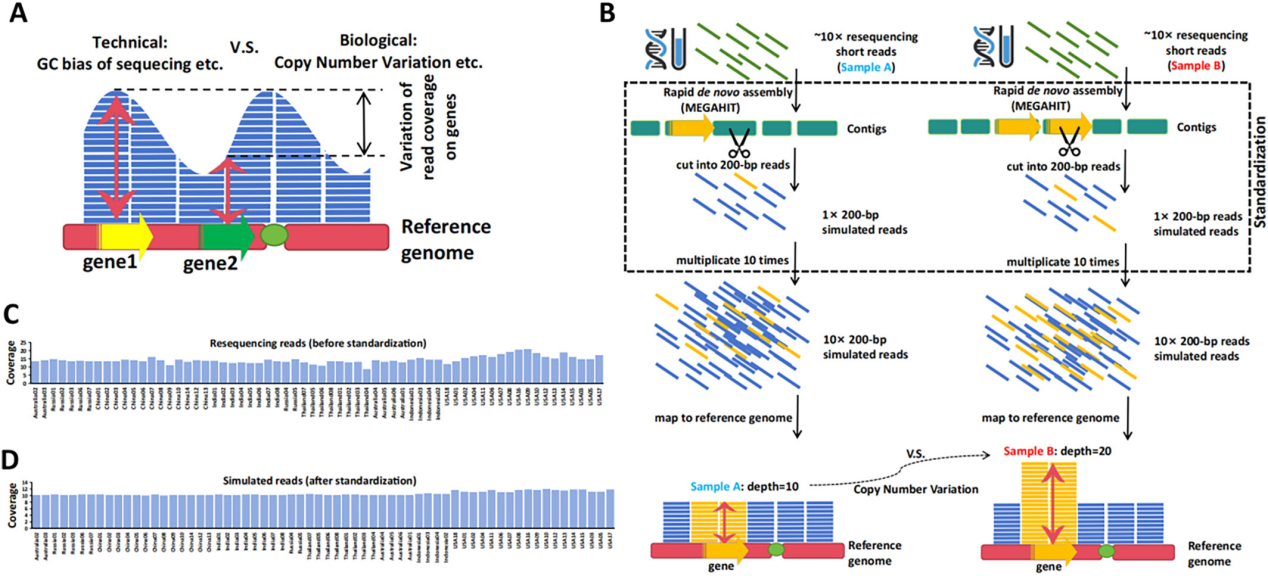
图1. ST4gCNV检测基因拷贝数变异流程。













